3. Filtrar pontos de ocorrência com faunabr
Source:vignettes/espacializando_faunabr.Rmd
espacializando_faunabr.RmdCarregando os dados
Antes de começar, utilize a função load_faunabr para carregar os dados. Para informações mais detalhadas sobre como obter e carregar os dados, consulte Primeiros passos com faunabr
library(faunabr)
library(terra)
#Carregar dados
bf <- load_faunabr(data_dir = my_dir, #Pasta onde foi salvo o arquivo com get_faunabr()
data_version = "latest", #Versão mais recente
type = "short") #Versão resumida
#> Loading version 1.3Aqui, vamos utilizar os dados baixados em português
(translate = FALSE em get_faunabr()).
Obter polígonos espaciais da distribuição de espécies
Fauna do Brasil fornece informações sobre os estados brasileiros e
países com ocorrências confirmadas das espécies. A função
get_spat_occ() extrai essas informações e retorna polígonos
espaciais (SpatVectors) que representam a distribuição da espécie.
Podemos escolher obter o SpatVector dos estados e/ou países.
#Espécies de exemplo
spp <- c("Panthera onca", "Mazama jucunda")
#Obter polígonos espaciais
spp_spt <- fauna_spat_occ(data = bf, species = spp, state = TRUE,
country = TRUE, verbose = TRUE)
#> Getting states of Panthera onca
#> Getting countries of Panthera onca
#> Getting states of Mazama jucunda
#> Getting countries of Mazama jucundaOs SpatVectors são armazenadas em uma lista aninhada por espécies:
par(mfrow = c(2, 2), mar = c(2, 0, 2, 0))
plot(spp_spt$`Panthera onca`$states,
main = paste0(names(spp_spt)[[1]], " - States"), mar = NA)
plot(spp_spt$`Panthera onca`$countries,
main = paste0(names(spp_spt)[[1]], " - Countries"), mar = NA)
plot(spp_spt$`Mazama jucunda`$states,
main = paste0(names(spp_spt)[[2]], " - States"), mar = NA)
plot(spp_spt$`Mazama jucunda`$countries,
main = paste0(names(spp_spt)[[2]], " - Countries"), mar = NA)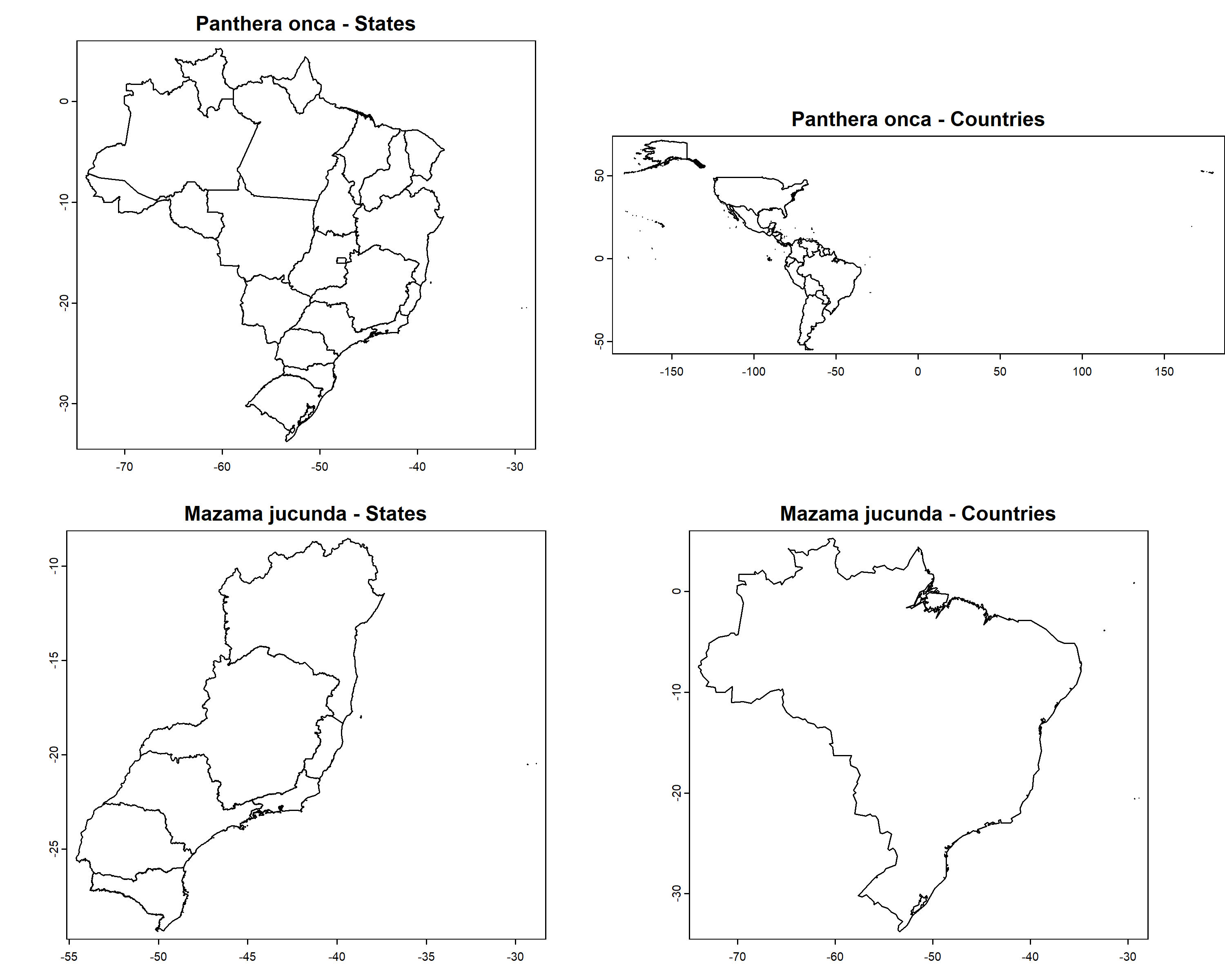
Filtrando registros de ocorrência usando informações de distribuição no Fauna do Brasil
Erros de georreferenciamento em registros de espécies online podem
introduzir vieses significativos nos resultados de pesquisas ecológicas
e biogeográficas. Alguns pacotes R, como o CoordinateCleaner,
ajudam a identificar erros comuns em dados de coleções biológicas, como
registros de organismos terrestres que caem no mar ou que foram
atribuídos a centroides de capitais e estados. Dado que as distribuições
de espécies no Fauna do Brasil são baseadas na expertise de
taxonomistas, elas representam informações valiosas para adicionar um
passo adicional na verificação da validade de registros de ocorrência
obtidos de bancos de dados online (como GBIF ou SpeciesLink). A função
filter_faunabr() automatiza essa identificação. Você pode
usar a função para sinalizar e/ou remover registros que estejam fora dos
estados brasileiros ou países com ocorrências confirmadas de acordo com
a Fauna do Brasil.
Como exemplo, vamos usar os registros de ocorrência de duas espécies.
Panthera onca é um felino terrestre com ocorrências confirmadas
em 22 dos 27 estados brasileiros (ausente no Rio Grande do Norte,
Paraíba, Pernambuco, Alagoas e Sergipe) e em 20 países. Chaetomys
subspinosus é um roedor arbóreo endêmico do Brasil com ocorrências
confirmadas em 5 estados (Bahia, Espírito Santo, Minas Gerais, Rio de
Janeiro e Sergipe). Vamos plotar mapas com essas informações usando a
função get_spat_occ():
my_spp <- c("Panthera onca", "Chaetomys subspinosus")
pol_spp <- fauna_spat_occ(data = bf, species = my_spp,
state = TRUE, country = TRUE,
verbose = TRUE)
par(mfrow = c(2, 2), mar = c(2, 0, 2, 0))
plot(pol_spp$`Panthera onca`$states,
main = paste0(names(pol_spp)[[1]], " - States"), mar = NA)
plot(pol_spp$`Panthera onca`$countries,
main = paste0(names(pol_spp)[[1]], " - Countries"), mar = NA)
plot(pol_spp$`Chaetomys subspinosus`$states,
main = paste0(names(pol_spp)[[2]], " - States"), mar = NA)
plot(pol_spp$`Chaetomys subspinosus`$countries,
main = paste0(names(pol_spp)[[2]], " - Countries"), mar = NA)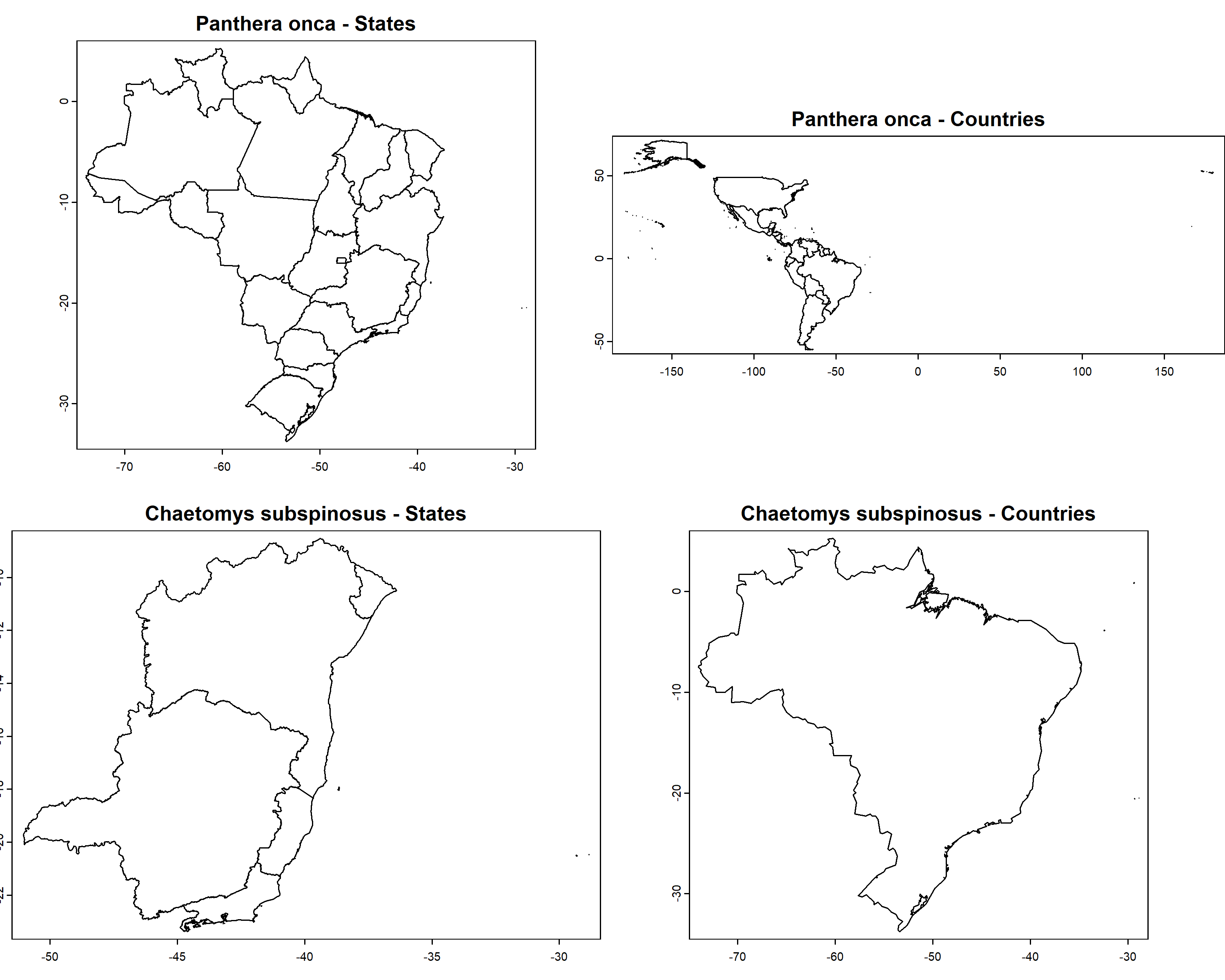 As ocorrências dessas
duas espécies foram baixadas do GBIF usando a função
As ocorrências dessas
duas espécies foram baixadas do GBIF usando a função
plantR::rgbif2(). Essas ocorrências estão salvas como
exemplos de dados no pacote (para mais detalhes, veja
?faunabr::occurrences). Podemos importá-las com:
data("occurrences")
head(occurrences)
#> species x y source
#> 1 Panthera onca -90.38409 17.377023 gbif
#> 2 Panthera onca -90.24368 17.240507 gbif
#> 3 Panthera onca -77.36680 0.287624 gbif
#> 4 Panthera onca -56.61023 -17.239688 gbif
#> 5 Panthera onca -61.04386 -2.387029 gbif
#> 6 Panthera onca -77.28850 0.288757 gbifOs dados de entrada com registros devem ser um dataframe com pelo menos 3 colunas: uma informando o nome da espécie, uma informando a longitude e outra informando a latitude. Vamos verificar se há registros fora das áreas naturais de ocorrência das espécies considerando estados e países:
occ_check <- filter_faunabr(data = bf, occ = occurrences,
by_state = TRUE, buffer_state = 20,
by_country = TRUE, buffer_country = 20,
value = "flag&clean", keep_columns = TRUE,
verbose = FALSE)
#> Returning list with flagged and cleaned occurrencesComo configuramos value = “flag&clean”, a função retornou uma lista com dois dataframes: um com todos os registros sinalizados se passaram (TRUE) ou não (FALSE) em cada teste; e outro apenas com os registros que passaram em todos os testes.
Vamos usar o pacote mapview para plotar um mapa interativo dos registros sinalizados:
#Instala pacote (se necessário)
if(!require(mapview)){
install.packages("mapview")
}
#Carregar pacote
library(mapview)
library(terra)Vamos verificar os registros de Panthera onca, plotando os registros e o mapa obtido anteriormente. Os pontos verdes representam os registros que passaram no teste dos estados, ou seja, registros dentro dos estados com ocorrência confirmada da espécie. Os pontos vermelhos são os registros que caem fora dos estados. Os pontos cinzas são registros que não foram testados porque estão fora do Brasil.
#Converter pontos para spatvector
panthera_occ <- subset(occ_check$flagged,
occ_check$flagged$species == "Panthera onca")
panthera_occ <- vect(panthera_occ, geom = c("x", "y"),
crs = crs(pol_spp$`Panthera onca`$states))
#Plot interativo
mapview(pol_spp$`Panthera onca`$states,
layer.name = "States with confirmed occurrences") +
mapview(panthera_occ, zcol = "inside_state", layer.name = "Inside state?",
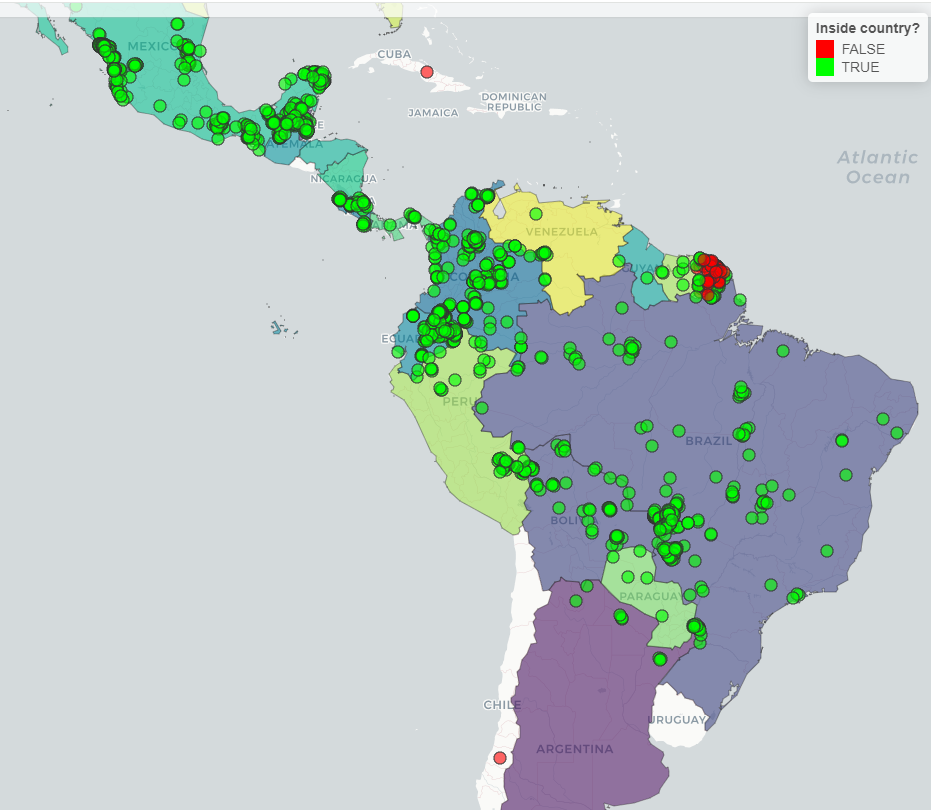
col.regions = c("red", "green"))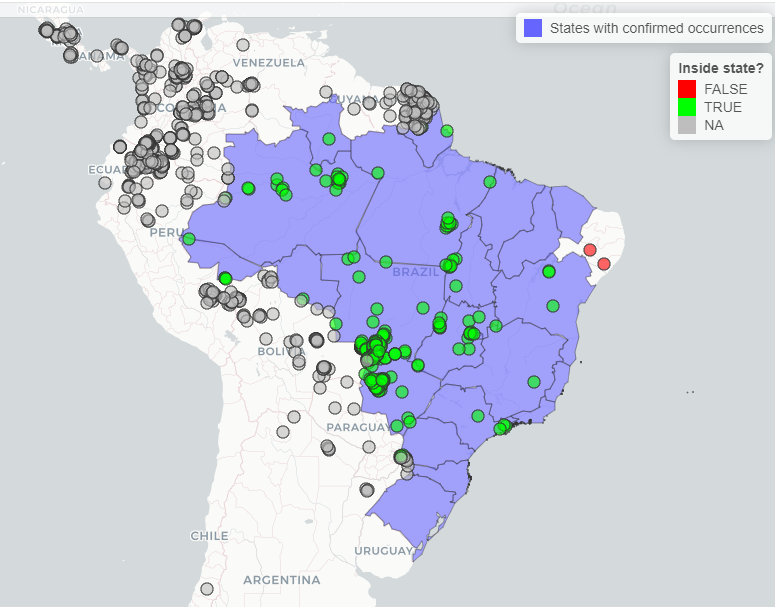 Podemos observar o mesmo com os países: os pontos verdes representam
registros que passaram no teste e os pontos vermelhos representam
registros que não passaram no teste.
Podemos observar o mesmo com os países: os pontos verdes representam
registros que passaram no teste e os pontos vermelhos representam
registros que não passaram no teste.
#Plot interativo
mapview(pol_spp$`Panthera onca`$countries,
layer.name = "Countries with confirmed occurrences", burst = TRUE, legend = F) +
mapview(panthera_occ, zcol = "inside_country",
col.regions = c("red", "green"),
layer.name = "Inside country?")