Uma forma de organizar dados de biodiversidade é usando matrizes de presença-ausência (PAMs), onde 1 representa a presença da espécie j na célula i, e 0 indica ausência. A partir de uma PAM, podemos estimar uma variedade de métricas relacionadas a padrões de biodiversidade, incluindo riqueza, tamanho da área de distribuição e composição. Para uma lista abrangente de métricas de biodiversidade, consulte a função PAM_indices do pacote biosurvey.
Carregando os dados
Antes de começar, utilize a função load_faunabr() para
carregar os dados. Para informações mais detalhadas sobre como obter e
carregar os dados, consulte Primeiros
passos com faunabr
library(faunabr)
#Carregar dados
bf <- load_faunabr(data_dir = my_dir, #Pasta onde foi salvo o arquivo com get_faunabr()
data_version = "latest", #Versão mais recente
type = "short") #Versão resumida
#> Loading version 1.3Aqui, vamos utilizar os dados baixados em português
(translate = FALSE em get_faunabr().
Obtendo uma matriz de presença-ausência
A função fauna_pam() facilita a utilização das
informações de distribuição de espécies no Fauna do Brasil para gerar
uma PAM. Cada local representa um estado brasileiro ou um país. Além da
PAM, a função também fornece um resumo e um SpatVector contendo o número
de espécies em cada local.
Como exemplo, vamos obter uma PAM com todas as espécies de mamíferos nativos do Brasil:
#Selecionar espécies de mamíferos nativos do Brasil:
br_mammals <- select_fauna(data = bf,
include_subspecies = FALSE, phylum = "all",
class = "Mammalia",
order = "all", family = "all",
genus = "all",
lifeForm = "all", filter_lifeForm = "in",
habitat = "all", filter_habitat = "in",
states = "all", filter_states = "in",
country = "BR", filter_country = "in",
origin = "all", taxonomicStatus = "valido")
#PAM em Estados do Brasil
pam_mammals <- fauna_pam(data = br_mammals, by_state = TRUE,
by_country = FALSE,
remove_empty_sites = TRUE,
return_richness_summary = TRUE,
return_spatial_richness = TRUE,
return_plot = TRUE)
#Visualizar (em tibble) PAM com as primeiras 5 espécies nos primeiros 5 sites
tibble::tibble(pam_mammals$PAM[1:7, 1:5])
#> # A tibble: 7 × 5
#> states `Platyrrhinus aurarius` `Kannabateomys amblyonyx` `Callicebus lucifer` `Cerradomys maracajuensis`
#> <fct> <dbl> <dbl> <dbl> <dbl>
#> 1 AM 1 0 1 0
#> 2 ES 0 1 0 0
#> 3 MG 0 1 0 1
#> 4 PR 0 1 0 0
#> 5 RJ 0 1 0 0
#> 6 RS 0 1 0 0
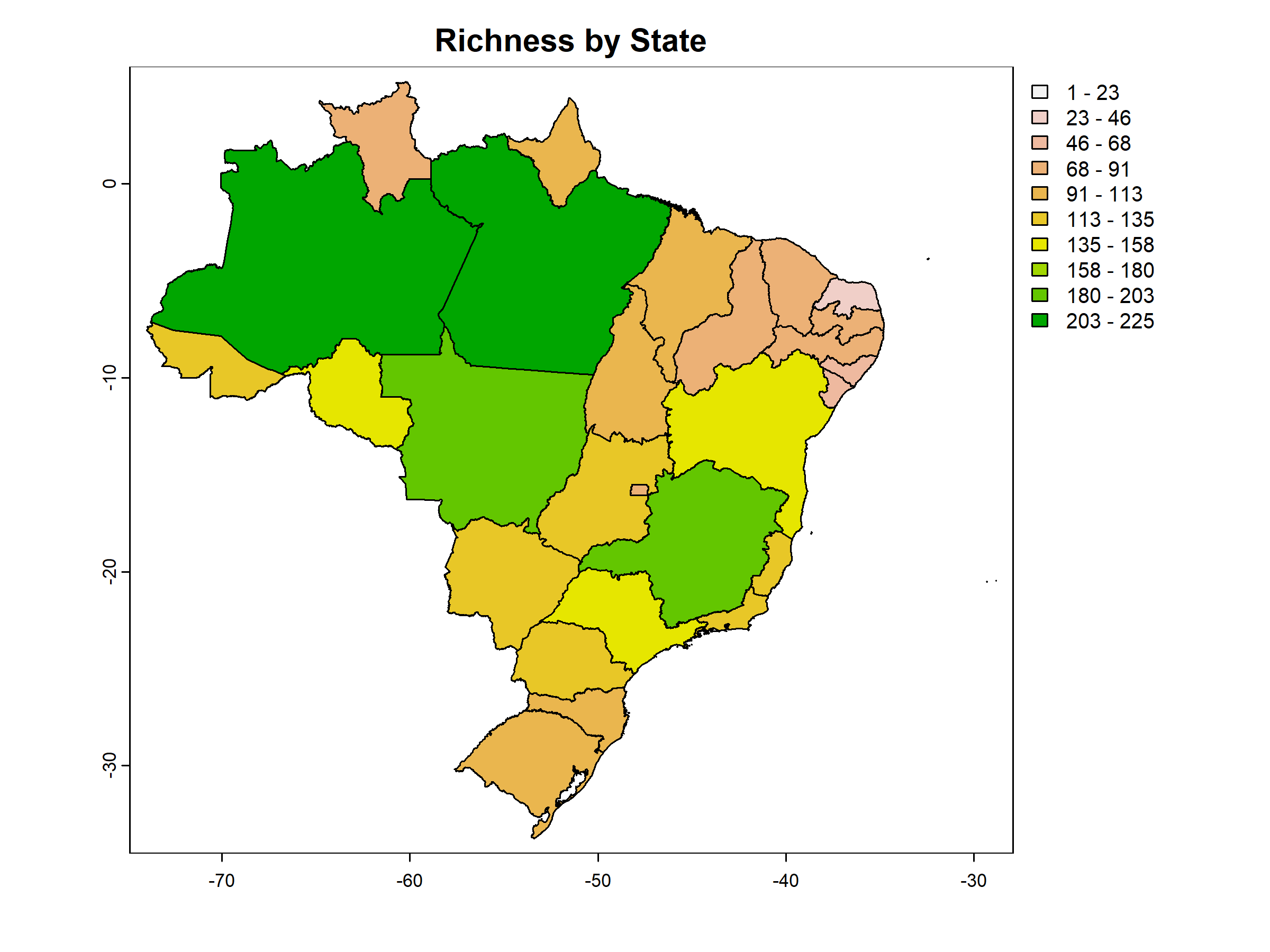
#> 7 SC 0 1 0 0Como return_richness_summary está definido como TRUE, a função também retorna um dataframe contendo o número de espécies por local.
#Visualise (em tibble) a tabela com a riqueza por site
tibble::tibble(pam_mammals$Richness_summary[1:7,])
#> # A tibble: 7 × 3
#> states richness
#> <fct> <dbl>
#> 1 AM 225
#> 2 ES 120
#> 3 MG 188
#> 4 PR 116
#> 5 RJ 133
#> 6 RS 105
#> 7 SC 101Se return_spatial_richness estiver definido como
TRUE, a função retornará um SpatVector contendo o número de
espécies por local. Além disso, quando return_plot também
estiver definido como TRUE a função retornará um plot do mapa: